第Ⅰ部 腸内細菌の基礎知識
1章 腸内細菌研究ことはじめ
福田真嗣
(慶應義塾大学先端生命科学研究所/順天堂大学大学院医学研究科/株式会社メタジェン/メタジェンセラピューティクス株式会社)
1腸内細菌の発見から20世紀前半まで
―便の観察からはじまった1)~3)
そもそも腸内に細菌がいると最初に報告されたのは1674年のことである(図1).オランダの科学者レーウェンフックが,自身が作成した顕微鏡でさまざまなサンプルを観察するなかで,小さな生物(ただし,生物であるかは当時は明らかでなかったかもしれない)が便に含まれるのを発見した.

その後パスツールが滅菌法を(1866年),コッホが純粋培養法を(1881年)それぞれ開発し,近代的な細菌学が開拓された.純粋培養法とは,1種類の菌のみが含まれるコロニーを培養する方法である.これらの技術を活用しながら,エシェリヒは1885年,乳児の便から大腸菌を発見した4).なお大腸菌の学名Escherichia coliのうち.Escherichiaは発見者であるエシェリヒの名前(Escherich)から,coliは発見場所である腸(colon)にそれぞれ由来している.まだこの頃には後述する嫌気培養法も確立されていなかったため,便を培養して生えてきたのは通性嫌気性菌(酸素のある環境でも増殖できる菌)であり,そのような環境でも生存できる大腸菌だった.
その後北里柴三郎らが破傷風菌の培養のために,酸素がない状態で培養する方法(嫌気培養法)を確立させた(1889年).彼が当時まだ特定されずにいた破傷風の原因菌の純粋培養を試みる過程で,試験管の底の方にだけ破傷風菌が増殖しているのを観察し「酸素がないほうが培養できるのでは」という着想から嫌気培養法を確立させたというエピソードがある5).
そして1899年,パスツール研究所のティシェが,腸内細菌の1つとしてビフィズス菌を発見した.ビフィズス菌という名前は顕微鏡で観察したときの,V字やY字に分岐した特徴的な形から,ラテン語で「二又の」を意味するビフィドゥス(bifidus)が語源となっている(Column❶※).翌年の1900年にはモローがアシドフィルス菌(Lactobacillus acidophilus)を発見した.このように,19世紀後半に腸内細菌の発見が相次いだ.
220世紀後半―腸内細菌の培養・同定・分類法の確立
1)光岡博士による包括的培養法の確立
1で紹介した嫌気培養法は,当時はまだ腸内細菌に特化したものではなかった.その後70年ほどを経て,東京大学の光岡知足博士らにより腸内細菌の包括的な培養法が開発された.光岡博士らは腸内細菌の培養に嫌気性培養を取り入れ,また培養基質(培地に含まれる栄養物質)も検討した結果,従来の10倍~100倍もの菌が生育したと報告している.なお,この手法により成人の便からビフィズス菌を単離したと発表するも,当時ビフィズス菌は乳児に特有の腸内細菌として認知されていたため,なかなか学会では認められなかったとのエピソードがある2).
ただし光岡博士による培養法により,すべての腸内細菌が培養できるようになったわけではない.残りの腸内細菌はいまだ培養法すらわからないという状態である.それらの培養が難しいのはなぜだろうか.その理由としては,腸内細菌の多くがやはり嫌気性細菌であるということと,それぞれの菌がどのような栄養を要求するのかがわからないことが大きい.ヒトゲノム配列がわかってもヒトのしくみの解明には程遠いのと同様,腸内細菌のゲノム配列が解読できるようになった今でも,タンパク質の機能の多くは不明であり,そのため培養条件を確立するための手がかりも少ない.腸内細菌は,4つの門(Bacteroidota,Bacillota,Actinomycetota,Pseudomonadota.旧名称はそれぞれBacteroidetes,Firmicutes,Actinobacteria,Proteobacteria)が主なものとして腸内に定着しているが,どのような栄養や環境条件を必要とするか,その多様性は大きい.したがって培養がうまくいかない腸内細菌もまだまだ多いのが現状であり,微生物学的な解析,生化学的な解析をもとにいろいろな条件を試していくのが現実的な方法となる.
とはいえ光岡博士により腸内細菌の培養法が確立されたことにより,ヒトや動物の腸内から,それまでと比べて多くの菌種,特に嫌気性細菌が新たに分離・同定された.すると腸内細菌叢を一種の生態系として捉えることが可能となり,多くの因子(食事,年齢,疾患,ストレス,薬物など)に伴う腸内細菌叢の変動が報告された1).なかでも,宿主の健康・疾患に腸内細菌叢が関与するという仮説が光岡博士により提唱され,現在の腸内細菌叢研究の礎となっている.
2)腸内細菌の分類法の確立
ところで,培養法が中心となり行われていた当時の腸内細菌の分類は,主に形態と生化学的な特徴(グラム染色などの染色法や,どういった栄養素により培養され,どのような有機酸が産生されるかなど)により行われていた.それが2000年代に入ると,遺伝子レベルでの解析と分類が行われるようになった.細菌がもっている16S rRNA(リボソームRNA)遺伝子の塩基配列をクローニングにより解析し,その類似度で分類する方法である.その後,遺伝子配列解析技術の発達により,ハイスループットに16S rRNA遺伝子を解析する手法(16Sアンプリコン解析,メタ16S解析などとよばれる)や腸内細菌叢ゲノムを網羅的に解析する手法(ショットガンメタゲノム解析)が登場し,現在主に用いられている(7章1参照).
分類に関しての詳細は2章1で解説しているが,分類学としては界門綱目科属種の分類があり(図2※),先ほど紹介したビフィズス菌は「Bifidobacterium属のlongum種」というように分類される.しかし私たち人間が一塩基多型(single nucleotide polymorphisms:SNPs)をもつのと同様,細菌でも種の下位に株(strain)という概念があり,株の違いにより菌の能力も変わってくる場合がある.例えばヨーグルトや乳酸菌飲料,整腸剤などのパッケージをご覧いただくと,株名までが記されているが,それも株により効能が異なることが想定されるからである.
3そして腸内マイクロバイオーム研究へ
1)オミクス研究の隆盛
21世紀に入り次世代シークエンサーや質量分析計などの解析装置が発展するに伴い,生物に存在する情報のすべてを解析するアプローチ(オミクス研究)が登場し,腸内細菌叢研究の発展にも大きく寄与している.本書で登場する「マイクロバイオーム」という言葉について解説する前に,ゲノムなどの用語について整理したい.
ゲノム(genome)という言葉は,遺伝子(gene)に,「すべて」を意味する「ome」をつけることで,遺伝子全体を指す言葉として生まれた.ゲノムという言葉に対する現在の意味合いは1930年に木原均博士により「ある生物をその生物たらしめるのに必須な遺伝情報」として概念的に定義された8).またその全体を解析する研究アプローチとしては「omics」が付くものとされ,遺伝子全体の場合はゲノミクス(genomics)となる.
同様にmRNAなどの転写物はtranscriptなのでその全体はトランスクリプトーム(transcriptome),タンパク質(protein)の全体はプロテオーム(proteome),代謝物質(metabolite)の全体はメタボローム(metabolome)となり,それらの研究アプローチはそれぞれトランスクリプトミクス(transcriptomics),プロテオミクス(proteomics),メタボロミクス(metabolomics)となる(表).
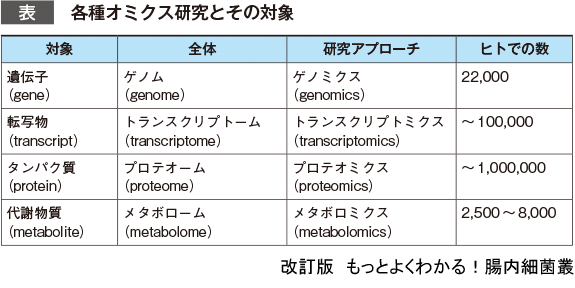
さて本書の主題となる微生物(microbes)に対して,その全体を指す言葉としてマイクロバイオーム(microbiome)がある.マイクロバイオームは,微生物集団が有する遺伝情報の全体を指す言葉として用いられる.一方,マイクロバイオータ(microbiota)という言葉もある.これは,日本語では微生物叢ともいうが,生きた微生物集団の全体を指す言葉として用いられる.そのため,次世代シークエンサーで解析された遺伝子情報はマイクロバイオームであり,腸内などでバランスが崩れるのは生きた微生物集団であるマイクロバイオータである.
腸内細菌(gut microbes)の場合は,その生き物全体を指すのが腸内細菌叢(gut microbiota)で,別名腸内フローラともよばれる.フローラという言葉はもともと生態学で叢(くさむら,多くのものが集まっている)を指す言葉として用いられていたもので,生物相などの「相」が転じて使われたのと,微生物が密集している様子が花畑(flora)に見えることから合わせて使われるようになった.
2)superorganismという概念
腸内には細菌がたくさん存在し,宿主であるわれわれ人間の細胞と相互作用している.具体的には,人間の細胞〔およそ30兆個(3.0×1013個)〕と腸内細菌〔およそ38兆個(3.8×1013個)〕9)が合わさって構成される.これは真核生物と原核生物という分類学上異なる生き物が1つの集団として存在している状態であるため,2000年にアメリカの分子生物学・微生物学者であるJoshua Lederberg博士はsuperorganism(日本語では超生命体,超個体などと訳される)という概念として提唱した10).思い返せば私たちヒトの細胞にもミトコンドリアという原核生物起源の細胞内小器官が存在している.本書でこれから紹介するように腸内細菌も私たちの体に多くの貢献をしてくれているため,腸内細菌もミトコンドリアと同じような共生の初期段階として捉えることができるかもしれない.

オミクスアプローチがもたらしたインパクトの大きさ
先述したとおりオミクスアプローチが腸内細菌叢研究に与えた影響は非常に大きい(オミクスアプローチについては7章で詳細を紹介する).ゲノミクスやトランスクリプトミクスと並行してバイオインフォマティクス(生物情報科学)が発達した.大量のデータを取得しそれらを解析できるようになる以前の研究の主流は,仮説検証型の研究,すなわちその研究分野の背景知識や研究者の経験から「このあたりが重要そうだ」と仮説を立てて検証する,というスタイルだった.しかしオミクス研究やバイオインフォマティクスの進展により,例えばゲノミクスであればヒトの遺伝子を網羅的に解析することが可能になった.その結果,例えば健康なヒトと病気のヒトのゲノムを調べ,病気の人はこの遺伝子に変異が入っている,というのを見つける方法〔ゲノムワイド関連解析(genome-wide association study:GWAS)〕が可能になった.このように,大量のデータをもとにそこから鍵因子を見つけるデータ駆動型の研究が可能になってきた.
たくさんの情報から答えに相当する情報を絞り込む過程に,バイオインフォマティクスが用いられる.この技術がないと,例えば20,000個の遺伝子情報を得た後に20,000個の遺伝子を目で見て1つずつ解析しなければならないが,そのサンプル数が数百検体あった場合,解析には途方もない時間がかかるだろう.オミクスアプローチの利点の1つは,遺伝子や転写物,タンパク質といった表の左列に相当する情報をすべて数値情報に変換し定量的かつ統計科学的に扱えることである.それによって例えば,この遺伝子に変異が入ったからこのタンパク質に影響が及びこの代謝物質の量が変化する,というように各階層の情報を関連付けながら解析する統合オミクスも可能になった.
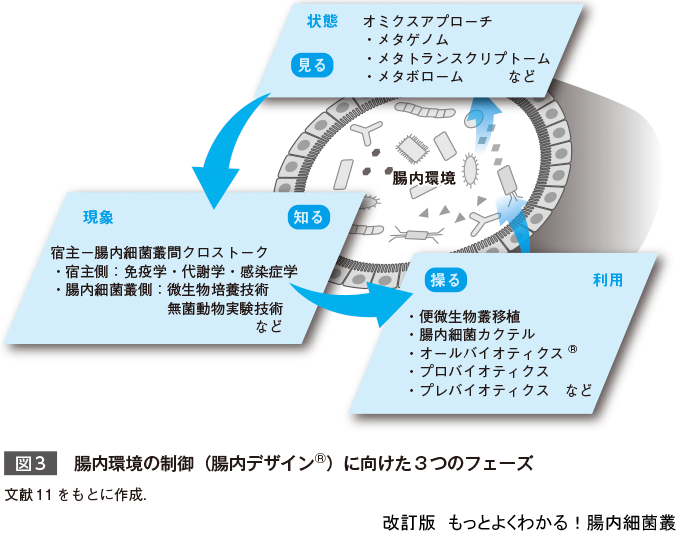
ただ,少なくとも腸内細菌叢研究分野においては,オミクスアプローチだけですべてがわかるというわけではないと筆者は考えている(図3)11).このようなアプローチにより得られたデータはあくまで,ある1つの状態を映し出す「スナップショット」でしかない.病気の人にはどのような菌が多い,どういった因子が多く発現しているという「相関関係」を見つけるのには適しているが,それが本当に病気の原因か結果かという「因果関係」まではわからない.腸内細菌叢を含む生命科学の分野において,病気を予防する,あるいは治療することをめざす場合,オミクスアプローチだけで完結することは難しく,そこで得た知見を動物実験などで評価しなければいけない.用いられる手法には細菌培養や無菌動物実験など従来の腸内細菌叢研究で培われた手法(8章参照),あるいは免疫学的,代謝学的アプローチなど他の学問分野の手法があり,腸内細菌叢の機能研究のためには,オミクスアプローチとあわせて行う必要があるだろう.
文献
- 光岡知足:腸内菌叢研究の歴史.実験医学,32:652-657,2014
- 「健康長寿のための食生活」(光岡知足/著),岩波書店,2002
- 「腸内細菌学」(光岡知足/編),朝倉書店,1990
- Escherich T:Fortschr Med, 3:515, 1885
- テルモ株式会社「医療の挑戦者たち30 破傷風菌の純粋培養」(2022年8月閲覧)
- 大野博司,中込 治:細菌の正しい日本語でのよび方.実験医学,30:3168-3169,2012
- 伊藤 研「分類ってなに?―『界・門・綱・目・科・属・種』の話―」(2022年8月閲覧)
- 武田洋幸「理学のキーワード:ゲノム」(2022年8月閲覧)
- Sender R, et al:PLoS Biol, 14:e1002533, 2016
- Lederberg J:Science, 288:287-293 2000
- 「明かされる“もう1つの臓器”腸内細菌叢を制御せよ!」(福田真嗣/企画),実験医学Vol.34 No.6,2016



