第2章 実験をはじめる前に検討すること
1 NGSを使った研究のはじめ方どこまで相談するか?
鹿島 誠1),小俣和輝2),加藤尚志3)
(東邦大学理学部生物分子科学科1),早稲田大学先進理工学研究科生命理工学専攻2),早稲田大学先進理工学研究科生命理工学専攻/早稲田大学教育・総合科学学術院3))
 次世代シークエンサー(next generation sequencer:NGS)は現代の生命科学においてなくてはならないツールになっている.しかし,分子実験から情報解析まで多岐にわたる知識と技能が必要とされるため,未経験の参入には大きな障壁があることも事実である.はじめてNGSを使ってみて期待した結果が得られなかった読者も多いのではないだろうか? そういった場合の多くは不適切な実験デザインに起因することが多い.本項では,経験者とのコラボレーションにより,実りあるNGSライフを送るためのポイントを紹介する.
次世代シークエンサー(next generation sequencer:NGS)は現代の生命科学においてなくてはならないツールになっている.しかし,分子実験から情報解析まで多岐にわたる知識と技能が必要とされるため,未経験の参入には大きな障壁があることも事実である.はじめてNGSを使ってみて期待した結果が得られなかった読者も多いのではないだろうか? そういった場合の多くは不適切な実験デザインに起因することが多い.本項では,経験者とのコラボレーションにより,実りあるNGSライフを送るためのポイントを紹介する.
はじめに
本項では,次世代シークエンサー(NGS)を “これから使おうとしている研究者” や “日常的にNGSを使用していない研究者” を対象に,NGSを利用しようと思ったときに,何を自分でやるべきか,何を人に頼むべきかに関して,Wet(組織実験・分子実験)とDry(データ解析)の両方を1人で行い,多数の共同研究を実施してきた筆者らの考えを紹介する.
先に結論を述べると,気軽に相談できる経験者とコラボレートして,実験デザインの段階から議論しながら誰が何をするのがよいのかを経験者と一緒に考えるという流れをおすすめする.共同研究をするとなると,自身で実験デザインを練り,何をお願いしたいかを決めてからでないと共同研究者にお願いしてはいけないと,特にNGS初心者は考えがちではないだろうか? これが危ないのである.なぜなのか,本項では筆者が考えるその理由を紹介し,どのような共同研究を行うのがよいのかを筆者らの共同研究の実例を交えて提案したい.
次世代シークエンサーを始めようとしてる研究者への注意点
1. NGSは一撃必殺技ではない
筆者(鹿島)が大学に入学した頃はすでにポストゲノム時代で,ゲノムがわかったのなら生物学にはもう謎は残っていないのではないかと思っていた.もちろんそんなことはないのだが,NGSの登場と普及により,1研究室レベルでもゲノムやトランスクリプトームが決定できるようになり,さらにはRNA-SeqやChIP-Seqなどの技術開発により2010年代は高い(当時は数サンプルで100万円規模の実験だった)けれどNGSを使えばなんでもわかるのではないかという空気が流れていた.例えば,「変異体のゲノムを読めば原因遺伝子はわかるはずだ」や,「RNA-Seqでコントロールと処理群を比較すれば原因遺伝子がわかるはずだ」という考えを筆者も抱いていた.今でもNGSを高価な「一撃必殺技」として思っている研究者は多いのではないだろうか?
しかし,NGSは研究者が求めているような明確な答えをもたらすのではなく,大量(数百~数千)の変異リスト・大量の発現変動遺伝子リストを生み出すのみである.むしろ,(語弊があるかもしれないが)気が付きたくない不都合な点も含めて網羅解析は真実をまるまる明らかにしてしまう.そのため,一撃必殺的にNGSを利用する研究では,大量の候補遺伝子のなかから解釈できる(研究者に都合のよい)遺伝子を探しだすことが生物学者の腕の見せどころになっていた.“最先端技術を利用した,知識と経験と勘を駆使した生物学”と言えば聞こえはいいが,当時の筆者は本当にこれでいいのかと常々感じていた.なぜなら,知識も経験もすでに既知の知見に基づいたものであり,研究者が真に追い求める未知の何かを発見しようとする方向性からは感覚的に逸れるからである.
そこで筆者が目をつけたのがデータ駆動型のアプローチである.簡単にいえば多検体を調べ,その共通項や差異と生命現象をみていくアプローチである.例えば,原因変異を探すゲノム解析の場合はゲノムワイド関連解析(genome wide association study:GWAS)が有効で,数百~数万検体のゲノム多型と形質情報の相関を調べれば,候補となる多型や変異を浮き彫りにできることはイメージできるのでないだろうか? 絞れなければ検体を増やせばいい.また,遺伝子発現解析の場合も数十規模で時系列や条件を設定することで,大量の発動遺伝子を時系列で整理し,より初期の変化を絞り込むことができる.絞り込みが足りなければ,発現変動遺伝子が検出されはじめる時点の前後をより細かく時系列RNA-Seqで調べればいい.
……理屈ではそうだが,そこまで大規模にやらないといけないのかというと,必ずしもそうではない.例えば,野生型と変異個体で形態的に差がなく,そのうえで本当に差異がないことを示したければ,おそらくn=3のRNA-Seqを一時点で行うだけで十分だろう.また,野外サンプルなどどうやっても数や条件数を用意できないケースもあるだろう.現実的には,実験目的と実験上の制約,コストと手間のバランスを統合して考えていく必要がある.めざすは実験目的を達成するうえで,できる限りコストと手間を減らすことであり,そのためには実際にNGSを利用したサイエンスの経験がある経験者から,失敗談・成功談を聞き一緒に実験計画を考えることが最善なのである.NGSは顕微鏡やPCRと同じで単なる1ツールであり,ただ使うだけで難問を解決してくれる「一撃必殺技」では決してない.一方でNGSの長短所をしっかり理解して“はまる使い方”をできれば,確かに強力な武器になる.その使い方はやはり使ったことがある人間に聞くのが最善であろう.
2. NGS未経験者との共同研究の進め方
多くの共同研究を行っていることと先に述べたが,ここでは筆者の典型的な共同研究の進め方を紹介する(図1).
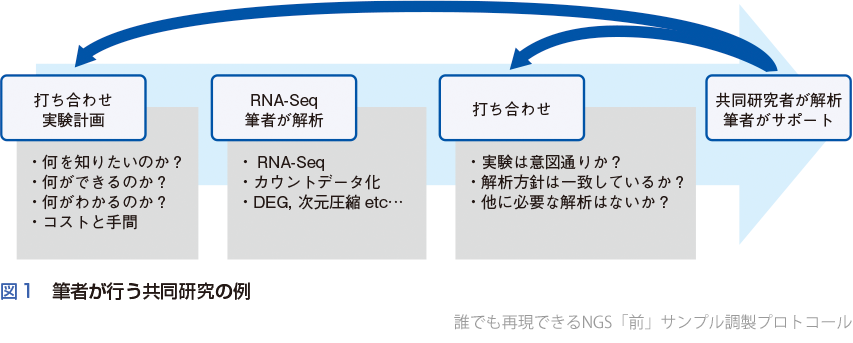
まず,(オンライン)ミーティングからはじまる.共同研究者からのプレゼンを通して研究の背景から課題までを聞き,「相手が何を知りたがっているのか?」,「何が壁になりサイエンスの進展を止めているのか?」を把握し,こちらからどのようなアプローチで何がわかるかもしれなくて,どれくらいのコストがかかるかを提案・説明し,両者の方向性のすり合わせを行う.そしてうまくすり合わせができた場合は共同研究がスタートになる.意外かもしれないが,注目している遺伝子の種類が少ない場合は,RNA-Seqではなく,qPCRや組織染色を筆者から勧め,とりあえず共同研究は保留というケースもそれなりにある.共同研究がスタートした場合は,Wet研究からDry解析までトータルで一緒にやっていくという流れだ.また,RNA-Seqは1回限りで終わらず,必要であれば初回の結果から次のサンプル条件を決めて再度RNA-Seqを行う流れを提案することもある.
以下では,実際の例として共著者の小俣氏に筆者との共同研究について紹介していただく.
共同研究の実例からみるNGSを用いた研究の落とし穴
1. RNA-Seqに至った経緯
筆者(小俣)は早稲田大学加藤尚志研究室の大学院生で,両生類の血液細胞における,ある生理活性分子の作用を調べている.種々の実験の結果,「何か作用しているんだろうけど明確な表現型がわからない」という状況にあり,網羅的解析ができれば明確な答えが得られるだろう,と淡い期待を抱いていた(図2).
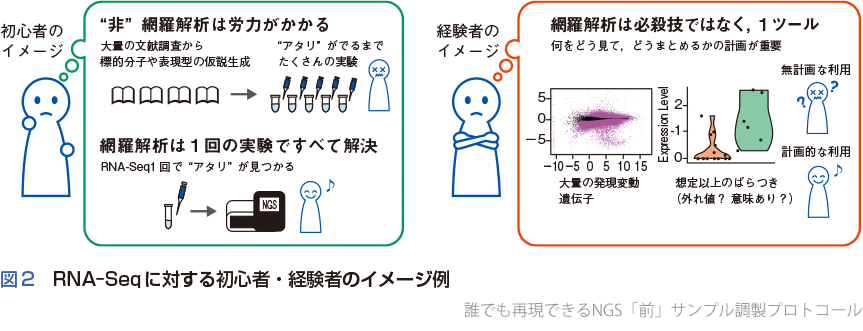
ちょうどそのような時期に,鹿島博士とのRNA-Seq解析の機会を得た.論文化途中のデータであるため仔細は記述できないが,以下では,実験デザインの打ち合わせから,結果を学会で発表するまでの経験を述べる.
2. 実験デザインの打ち合わせでの議論は重要
打ち合わせでは,これまでのデータを踏まえてどのようなことを期待しているかをプレゼンし,RNA-Seqの実験デザインを鹿島博士に相談した.当初,筆者は細胞の生理活性分子に対する応答を調べたかったため,n=3で刺激あり/なしで培養後少し経った後の計6サンプルという最小セットをとろうと考えていた.「初心者としてはできるだけシンプルなデータと向き合えた方が解釈しやすくて嬉しい」と思いつつの提案であった.しかしここで鹿島博士から計画変更の提案が3つ入った.
1つ目は,動物の体から採取して培養系にもち込んだ細胞(初代培養細胞)では,培養による影響でトランスクリプトームが変化する可能性があるため,採取直後の群を設定するべきという提案だった.動物の体内と人工的な培養条件では,細胞が異なる刺激を受けており,アーティファクトな影響を知るために必要だろうという内容だった.
2つ目は,時系列を見るべきという提案だった.仮にタイムポイント1点のみでRNA-Seqを実施し,残念ながら目的の変化(例えば発現変動遺伝子)を検出できなかった場合を考えてみよう.次に何をやるかの第一選択はもちろん,実験条件を変えて試料を調製し,再びRNA-Seqを実施することだろう.しかし,変えるべき条件は細胞採取方法か,培地か,どこから変えればよいものか判別は難しい.その際,最低限の2点だけでも時系列があることで,遺伝子発現のタイムコースを推定することができる.これによって,培養時間を後ろ/前にずらせば変化が見えそうだとか,変えるべきは時間ではないだとか,再実験にあたってのヒントが得られる.
3つ目は,試料調製の条件は公的リポジトリに登録されているカウンターデータに近づけるとよい,という提案だった.そうすることで,公的リポジトリ(例えば,NCBIのGene Expression Omnibus:GEO)に登録されたデータと自分のRNA-Seqの結果を比較することができる.もちろん,異なる実験系でのデータを比較する以上,比較結果の解釈には制約があるが,できるだけ条件が似ている方が議論しやすいだろう.比較の結果,「自分の結果の再現性確認」だけでなく,「同じホルモンでも細胞の種類が違うと応答が違う」といった深い解釈につながる可能性がある.
このように,自分自身でRNA-Seqをした経験がなければとても思いつかない提案がいただけるため,実験デザインを熟練者に相談するのは必須である.
3. データが出る前にやっておけることを確認しよう
「今までRT-PCRでチマチマ調べてた遺伝子発現が,これからはExcelで遺伝子名を検索するだけでわかるのか~」とワクワクしながら結果が出てくるのを待つあいだ,鹿島博士からのアドバイスを受けて,それまでの実験で活性化が確認できていた転写因子の標的遺伝子や血液細胞に特異的な遺伝子のリストを準備した.満を持してそれらとRNA-Seqデータとをつきあわせると,トランスクリプトーム全体ではきちんと血球で発現している遺伝子をとらえることができていた.さらに,既知の転写因子の下流遺伝子も発現変動遺伝子(differentially expressed genes:DEGs)として検出することができており,胸をほっとなでおろした.筆者の研究は,ここからさらに「この細胞で何かが起きているのか」をさぐる探索的な研究であったため,前述のような安心材料があることで,地に足の着いた気持ちでその後の解析を行うことができた.自身のデータが真実を反映しているかの疑義をもつことなく,安心してデータを扱うためにも,解析結果が出るまでの期間に注目している遺伝子のリストを準備することは重要である.
筆者の場合は参考文献が豊富に手元にあり,リストをすぐに準備できたが,テーマをはじめたての場合はそうもいかないだろう.そういった場合,まず総説を参照することは有効である.加えて,近年では,遺伝子の機能を体系的に整理したデータベースが整備されているので,そこに情報がないか探してみるのもよいだろう.
例えば,遺伝子オントロジー(gene ontology:GO)から,求める遺伝子リストを探してダウンロードするのがラクである.具体的な手順は,まず,遺伝子オントロジーのWebサイトにアクセスしたら,“Search GO term” から関連のある単語(血液細胞ならhematopoietic cellなど)を調べ,出てくるGO termから最も関連のあるものを選択し,ページ内のダウンロードから遺伝子リストを取得する.これで完了である.なお,生物種やGO termによって,登録されている情報量に隔たりがあること,種によって遺伝子名が違うことに注意されたい.
4. データ解析の紆余曲折も相談しよう
RNA-Seq解析では,めでたくある程度のDEGsを検出することができた.しかし,DEGsをGOエンリッチメント解析にかけると,それらしい生物学的プロセスはヒットしなかった.すなわち,当初もくろんでいた一撃必殺的な結果はでなかったのである.「DEGsがあるのに,なぜGOが拾えないのだろう……」「これでは論文どころか学会発表のネタにもできない……」とモヤモヤした.この結果を研究室内で議論したところ,「GOではなく,パスウェイ解析に回してはどうか」という意見があり,実施することになった.パスウェイ解析はエンリッチメント解析である点においてはGO解析と近いが,対象となるtermが異なる.このパスウェイ解析の結果,GOエンリッチメント解析ではヒットしなかったパスウェイがヒットし,筆者の実験系で細胞がどのような応答をしているかのヒントが得られた.しかし,「これなら学会で発表できるぞ!」と喜び,鹿島博士に結果を報告したところ,以下のような解析の問題点を指摘された.
筆者が使用したパスウェイ解析では,入力される遺伝子データが哺乳類ゲノムをバックグラウンドとすることを前提としている.しかし,哺乳類と両生類とでは,細胞分裂や解糖などの普遍的な生命現象であっても,それらにかかわる遺伝子の数は違うかもしれない.すなわち,筆者が行った統計検定の前提が間違っていたのである.最終的に,「もし学会で発表するなら,最低限,“両生類で得られたDEGsをマウスのオルソログに変換してマウスのパスウェイ解析にかけた” という書き方をするべきだ」というアドバイスをいただき,なんとか発表にもっていくことができた.
共同研究者に相談していなければ,誤った解析手法に気づくことができず,間違った仮説にもとづいた発表・実験につなげていたかもしれない.もしくは,論文を投稿してから査読者に指摘されて損をしていたかもしれない.オミクス実験に慣れていないと思わぬ間違いをおかすことがあるため,読者の方々には,実験デザインの段階で失敗が明らかにできたり,発表する前に解析の誤りに気づけたりするよう,共同研究者への相談を恐れずにしていただけると幸いである.
最後に
後半の小俣氏のケースは共同研究者に恵まれたケースだと思われるかもしれない.確かに日々多くの案件と向きあっている共同研究者は,多忙に見え,相談をためらうこともあるかもしれない.しかし,最低限のマナーを守っているのであれば相談して悪いことはないはずだ.もちろん,共同研究者もキャパシティーの問題で対応できる範囲は決まってくるかもしれないが,その共同研究テーマが面白いのであれば,きっと最大限力を貸してくれるはずだ.なので,Wetをメインに研究している方は,遠慮せずDry研究者と話していくことを強くお勧めする.



